XIII. LOS VIRUS Y LA INGENIERĶA GENÉTICA
UN FENÓMENO comúnmente observado en el laboratorio consiste en
que un fago capaz de propagarse en una cepa bacteriana determinada se replica
en forma ineficiente cuando es crecido en otra cepa diferente. Se dice que estos
fagos están restringidos cuando se encuentran en una cepa bacteriana diferente
de aquella en la cual han sido propagados originalmente. Sin embargo, casi siempre
una pequeña proporción del fago es capaz de evadir el estado de restricción
y crecer de manera adecuada en la nueva cepa bacteriana, pero la progenie de
este fago será incapaz de crecer en la cepa bacteriana que originalmente permitió
la propagación del fago precursor. Estas observaciones sugieren que una modificación
específica inducida por la nueva bacteria hospedera protege al fago de manera
que no puede ser restringido dentro de la nueva cepa bacteriana, pero a la vez
pierde la capacidad para crecer en la antigua cepa hospedera.
A principios de los años sesenta, Bertani, Weigle y Arber demostraron en forma
independiente que la modificación inducida por el hospedero ocurre en el nivel
del ADN del fago, y el fenómeno de restricción es consecuencia
de la degradación por hidrólisis enzimática del ADN viral que no
ha sido modificado. El ADN de la bacteria hospedera y otros
ADNs presentes en dicha céla son modificados por la adición de grupos
metilo (CH3) en sitios específicos
los cuales son normalmente reconocidos por un tipo de enzimas conocidas como
enzimas de restricción, las cuales solamente pueden degradar ADN
no metilado. Así, la metilación de una base en particular presente en la secuencia
de nucleótidos reconocida por la enzima, impide la hidrólisis y ruptura del
ADN en la región de esta secuencia. Las enzimas de restricción
son endonucleasas capaces de reconocer secuencias específicas de nucleótidos
en el ADN de cadena doble y cortar ambas cadenas a la altura de
dichas secuencias. Cada tipo de enzima de restricción reconoce una secuencia
de nucleótidos en particular, la cual generalmente consta de cuatro a seis pares
de bases. Una caracterísuca notable de los sitios donde actúan las enzimas de
restricción consiste en que la secuencia de bases es palindrómica y el sitio
de corte está localizado en forma simétrica con respecto al doble eje de simetría,
por ejemplo: la secuencia reconocida por la enzima de restricción Eco Rl,
obtenida de la bacteria E. coli, es:
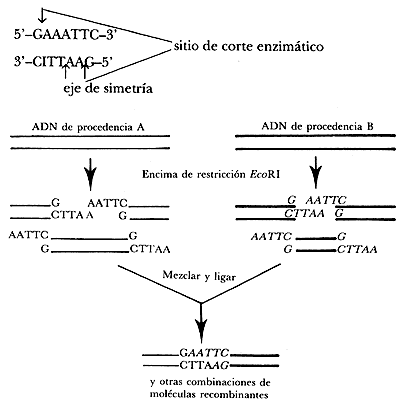
FIGURA XIII.1. Recombinación in vitro de dos fragmentos
de ADN.
En este caso, la unión AG presente en cada cadena de nucleótidos es hidrolizada por la endonucleasa.
Hasta la fecha han sido purificadas y caracterizadas más de cien diferentes enzimas de restricción. Su nomenclatura consiste en una abreviatura de tres letras correspondientes a la bacteria productora de la enzima (por ej.: Hae = Haemophilus aegyptum) más una letra en ciertos casos, que designa a la cepa bacteriana, y un número romano, por ej.: Hae III.
La caracterización de las enzimas de restricción ha permitido desarrollar técnicas
refinadas para manipular los genes. El factor principal en estas técnicas está
dado por la capacidad de ciertas enzimas de restricción para producir cortes
escalonados (indentados) en sitios bien definidos presentes en las moléculas
de ADN. La figura XIII.1 ilustra la manera en que estas enzimas
pueden ser utilizadas: ADN procedente de dos fuentes diferentes
es cortado con la misma endonucleasa (por ej.: Eco Rl) para producir
fragmentos que poseen colas o términos de cadena sencilla cuyas secuencias de
nucleótidos son complementarias. Incubando mezclas de ambos fragmentos en condiciones
que favorecen la reunión de los mismos en presencia de la enzima ADN
ligasa, es posible producir fragmentos de ADN híbrido también conocido
como ADN recombinante.
Es relativamente sencillo introducir en una bacteria fragmentos de ADN
por medio del proceso conocido como transformación. Sin embargo, dichos fragmentos
de ADN no pueden replicarse y acaban siendo eliminados. Para evitar
este problema, el fragmento de ADN es insertado en un vector
o vehículo de clonación, el cual consiste en una molécula de ADN
capaz de replicarse en forma autónoma. Entre los vectores más a menudo utilizados
se encuentran los plásmidos bacterianos, que son pequeñas moléculas de ADN
circular de cadena doble, los cuales generalmente contienen genes que codifican
toxinas o enzimas capaces de inactivar ciertos antibióticos. Los plásmidos son
cromosomas bacterianos accesorios y se diferencian del cromosoma principal en
que los plásmidos no son estrictamente necesarios para la subsistencia y reproducción
de la bacteria en cuestión. Los plásmidos pueden replicarse independientemente
del cromosoma principal. Las bacterias pueden contener plásmidos tipo unicopia
o multicopia (más de 20). En general, los plásmidos multicopia son pequeños
y a menudo se utilizan como vehículos moleculares de donación. Por ejemplo,
una célula de E. coli generalmente contiene 20 moléculas (copias) de
plásmidos y sólo una molécula del cromosoma principal.
Otros vectores de donación están representados por ciertos bacteriófagos, particularmente
el fago ha sido utilizado con éxito en diversos protocolos de manipulación genética.
El fago l tiene dos grandes ventajas como
vector de clonación. En primer lugar, el ADN recombinante puede
ser preparado en grandes cantidades, ya que cada bacteria produce cientos de
copias del ADN viral recombinante. Dicho ADN recombinante
puede ser purificado fácilmente, ya que aparece como componente de las nuevas
partículas del fago l . Utilizando cepas mutantes
del fago l defectuosas en su capacidad para
lisar la célula hospedera, es posible incrementar el rendimiento de nuevas partículas
virales, las cuales pueden ser recuperadas induciendo artificialmente la lisis
de la bacteria hospedera. El actual conocimiento detallado de la estructura
del genoma del fago l permite construir mutaciones
que incrementan la transcripción del ADN recombinante insertado
en el genoma del fago l. Es posible eliminar
grandes regiones del genoma del fago l que
no son esenciales para la replicación del ADN viral y la lisis
de la bacteria hospedera; las regiones eliminadas pueden ser substituidas por
fragmentos de ADN recombinante procedente de las más variadas fuentes.
Solamente se requiere un 25% del genoma del fago l
para permitir el crecimiento lítico de este fago en la bacteria hospedera. Sin
embargo, la eliminación del exceso de ADN viral, incluso de ADN
que no contiene secuencias esenciales para la replicación del fago provoca que
las nuevas copias de ADN no pueden ser empacadas en partículas
virales. Esta última propiedad es muy útil en ingeniería genética, pues el ADN
del fago l puede ser reducido en tamaño cortándolo
con una enzima de restricción posteriormente, este ADN
l puede ser mezclado con un ADN foráneo que ha sido
cortado con la misma enzima de restricción, de manera que ambos tipos de ADN
pueden recombinarse formando un ADN híbrido de suficiente tamaño
como para ser introducido por transfección en bacterias susceptibles. El ADN
recombinante puede replicarse en la bacteria, dando origen a nuevas moléculas
de ADN recombinante con un tamaño suficiente para ser empacadas
en partículas virales. Sólo las partículas que contienen ADN recombinante
en cantidad equivalente a 7S-105% del ADN originalmente presente
en el genoma del fago l pueden ser empacadas
en nuevas partículas virales que producirán la formación de placas en un cultivo
infectado; de esta manera, pueden ser distinguidas y separadas de aquellas partículas
que contienen un ADN l de tamaño
insuficiente debido a la ausencia de recombinación con el ADN foráneo
(figura XIII.2).
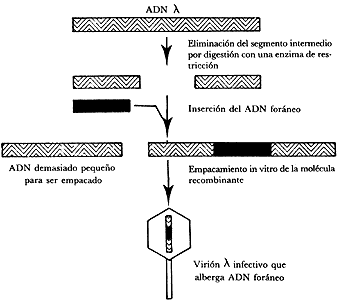
FIGURA XIII.2. Esquema que muestra cómo puede ser utilizado
un mutante del fago 1 como vector de clonación. La reacción de empacamiento
selecciona las moléculas de ADN recombinante.
Actualmente existen métodos análogos a los empleados para donar ADN
recombinante en bacterias, para propagar fragmentos de ADN recombinante
en células eucarióticas. En este caso, los vectores de donación están representados
por virus de animales como el SV4O, los adenovirus y algunos rotavirus, además
de algunos virus de plantas útiles para clonar genes en células vegetales.