III. MIRANDO DENTRO DEL GENE
LOS organismos vivos están caracterizados desde el punto de vista funcional por su capacidad para automantenerse y autorreproducirse. Existen tres tipos de moléculas gigantes o macromoléculas que normalmente son sintetizadas sólo en los organismos vivos y que son básicas para llevar a cabo estas funciones. Cada una de estas macromoléculas consiste de una larga cadena compuesta de muchas unidades estructurales. Estas pequeñas unidades discretas o monómeros van uniéndose unas a otras hasta formar un dímero (dos unidades), un trímero (tres unidades), etc., hasta formar un polímero. Las tres clases de macromoléculas o polímeros son: los polisacáridos, los polipéptidos y los polinucleótidos.
Los polisacáridos tienen monómeros o azúcares que contienen carbono (C), hidrógeno (H) y oxígeno (O) como la glucosa, fructuosa o galactosa. Los polipéptidos están formados de aminoácidos que contienen C, O, H, N (nitrógeno) y algunas veces azufre (S). Existen 20 diferentes clases de aminoácidos en los organismos. La unión entre dos aminoácidos se hace por medio de un enlace peptídico para producir un dipéptido. Una proteína está compuesta de una o varias cadenas de polipéptidos.
Por último, los polinucleótidos, también llamados ácidos nucleicos, pueden ser de dos clases: los polirribonucleótidos o ácidos ribonucleicos (ARN) y los polidesoxirribonucleótidos o ácidos desoxirribonucleicos (ADN). Los monómeros que forman los ácidos nucleicos están constituidos por una base, un azúcar y un fosfato, cuyos componentes químicos son C, O, N, H y P (fósforo). Este tipo de macromoléculas, los ácidos nucleicos, contienen la información necesaria para la replicación de los seres vivos, por lo que son el material genético presente en todo tipo de organismos. Como ya mencionamos el material genético de algunos virus puede ser ARN o ADN, y que el material genético de los organismos celulares es ADN.
Químicamente el ADN consiste de un par de cadenas que semejan
los ejes de una escalera; cada cadena tiene un esqueleto de fosfatos y azúcares
alternantes. Estos azúcares son de una sola clase, desoxirribosa (recordemos
que será ribosa para el ARN), compuestos de C, H y O en donde cuatro
de sus cinco átomos de C están formando un anillo con un átomo de O (Figura.
12).
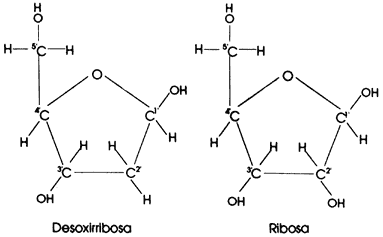
Figura 12. Desoxirribosa y ribosa.
A cada azúcar está ligada una base orgánica. Esta base está compuesta
de C, H, O y N, y puede ser de cuatro tipos: adenina (A), -timina (T) uracilo
(U) para el ARN-, citosina (C) o guanina (G). La citosina y la
timina son pirimidinas las cuales contienen dos N y cuatro C formando
un anillo. La adenina y la guanina son purinas con cuatro N y cinco C
arreglados en dos anillos (Figura 13).
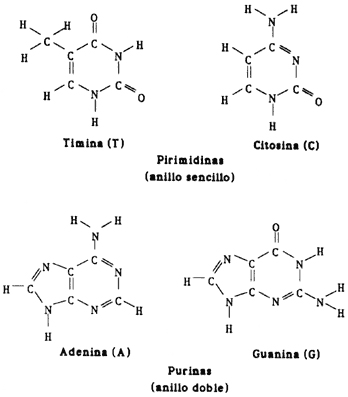
Figura 13. Bases nitrogenadas: purinas y pirimidinas.
Cada base orgánica de cada cadena se une a la otra base de la otra cadena mediante
un enlace o puente de hidrógeno: G y C se unen mediante tres enlaces de hidrógeno
y A y T mediante dos (Figura 14).
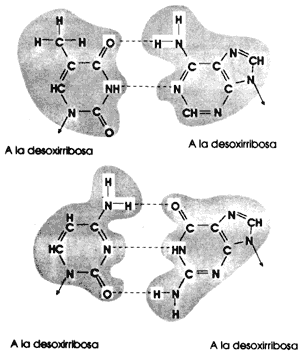
Figura 14. Figura que muestra las cuatro bases, tiamina, adenina, citosina
y guanina. El apareamiento de bases se da entre A y T y entre G y C, unidas
por puentes de hidrógeno (líneas punteadas); dos puentes entre tiamina y adenina,
y tres entre citosa y guanina.
Como ya hemos mencionado, el ADN es una doble hélice y sus características
principales están determinadas por los azúcares que se orientan en una dirección
en una cadena y en otra dirección en la otra cadena (Figura 15). Debido a este
arreglo inverso de los azúcares en cada cadena el ADN gira una
vuelta completa (es decir, 360 grados) cada 10 pares de bases (Figura 16).
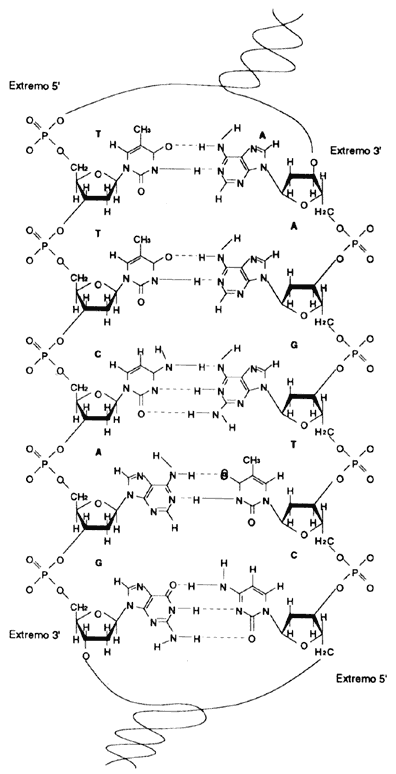
Figura 15. Molécula de ADN. Polímero doble de ADN,
en donde una hebra aparece de cabeza en relación a la otra. Cada polímero está
formado por nucleótidos unidos covalentemente a través del azúcar-fosfato; las
dos hebras están unidas entre sí por puentes de hidrógeno entre las bases adyacentes.
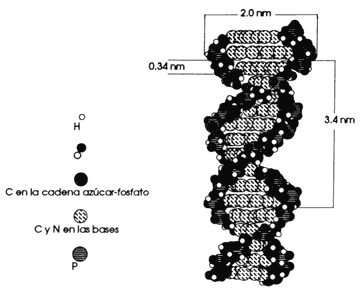
Figura 16. Modelo de la doble hélice de ADN. Las medidas fueron determinadas mediante estudios de difracción de rayos X. Cada par de bases tiene 0.34 nm de espesor, y diez pares producen una vuelta completa de la hélice, con una longitud de 3.4 nm. El ancho total de la doble hélice, incluyendo el par de bases y el esqueleto de azúcar-fosfato es de 2.0 nm.
Otra característica importante es que al esqueleto de azúcar-fosfato puede unirse cualquier base, púrica o pirimídica, teniéndose teóricamente, cualquier arreglo o secuencia de ellas. Pero, las bases de una cadena deben complementarse con las bases de la otra cadena; ya hemos mencionado que G sólo se une con C y A sólo lo hace con T. En otras palabras, A se complementa con T, y G se complementa con C, de tal suerte que si nosotros sabemos la secuencia de bases en una cadena podremos deducir la secuencia de la cadena opuesta. De esta forma, en una cadena doble de ADN el número de As es igual al número de Ts, y el número de Gs es igual al número de Cs. Por ejemplo, si sabemos que una secuencia de una determinada región de una cadena de ADN es ATTGC podremos deducir que la cadena opuesta tendrá la secuencia TAACG para esa misma región. Y es así como están constituidos los genes: trozos de ADN cuya secuencia es determinada y distinta de otros genes.
El descubrimiento de que el ADN es la molécula de la herencia es relativamente joven pues pertenece al siglo XX, y sin lugar a dudas ha sido uno de los hallazgos más sobresalientes de la biología.
¿Cuál es el material hereditario? Fueron muchos los experimentos diseñados y las hipótesis propuestas para contestar esta pregunta: Mencionaremos las aportaciones más importantes que marcaron el camino para dilucidar la estructura del ADN.
En 1928, el bacteriólogo Fred Griffith estaba interesado en la virulencia (capacidad de infectar y producir enfermedad) de las bacterias causantes de la neumonía, llamadas Pneumonococcus. Primero obtuvo dos cepas, una infecciosa y otra no infecciosa o inofensiva. La diferencia principal entre estas dos cepas era que la cepa virulenta o mortal sintetizaba una cubierta lisa de polisacárido que protegía a la bacteria de ser digerida por el hospedero (el organismo al cual infectan), mientras que la cepa inofensiva no podía sintetizar la capa protectora (y por eso era atacada por las defensas del hospedero, haciéndola efectivamente inofensiva); al ponerlas a crecer en una caja de Petri bajo cultivo, Griffith notó que la cepa virulenta formaba placas lisas, mientras que la inofensiva producía placas rugosas.
Como primer paso, Griffith inyectó a ratones normales con estas dos cepas para ver qué sucedía. El resultado fue poco sorprendente. Los ratones que fueron inyectados con la cepa lisa o virulenta murieron, mientras que los que recibieron la cepa rugosa, sobrevivieron. El segundo paso fue aplicar calor a las bacterias de la cepa lisa o virulenta para matarlas (como sabemos, la mayoría de las bacterias mueren con el calor, de ahí que, por ejemplo, debamos hervir el agua antes de tomarla para garantizar la muerte de estos microorganismos), e inyectarlas posteriormente a los ratones. Los ratones no murieron. Recordemos que la diferencia entre las dos cepas es la presencia de una capa de polisacárido protectora. Fue así como Griffith demostró que el polisacárido por sí solo no infectaba a las células de los ratones cuando la bacteria estaba muerta.
Hasta aquí todo había salido como Griffith lo esperaba. Pero el siguiente paso fue lo más importante. Se le ocurrió mezclar bacterias muertas de la cepa virulenta lisas (a las cuales les había aplicado calor) con bacterias vivas de la cepa inofensiva, y las inyectó en los ratones. ¿Qué creen que pasó? Pues simplemente que los ratones murieron de neumonía. ¿Cómo explicar esto, si las bacterias virulentas estaban muertas? Al hacer las autopsias de los ratones Griffith encontró bacterias lisas vivas. ¿De dónde salieron?
Lo primero que pensó Griffith fue que había cometido algún error, en algún
punto y que no había matado a las bacterias lisas, o que había habido contaminación
y por lo tanto accidentalmente había inyectado bacterias virulentas vivas en
los ratones, produciendo su muerte. Griffith procedió a repetir varias veces
su experimento. El resultado fue el mismo: los ratones murieron al serles inyectada
una mezcla de bacterias lisas muertas con bacterias rugosas vivas (Figura 17).
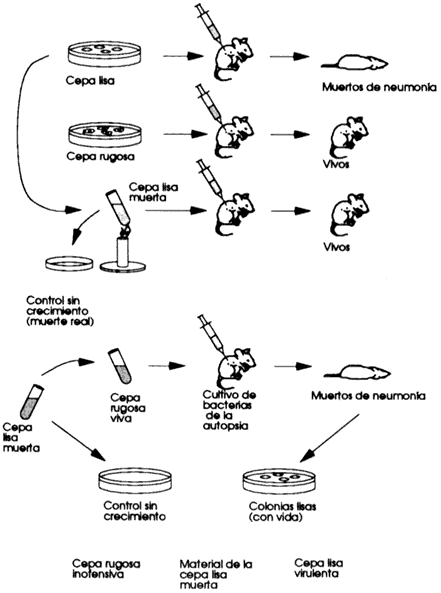
Figura 17. Los experimentos de Griffith contribuyeron a sustentar la idea
de que el ADN era el material genético. En su experimento, Griffith
trabajó con dos cepas de pneumonococcus (agente causante de la neumonía
bacteriana). La cepa lisa era virulenta mientras que la cepa rugosa era inocua.
Griffith intuyó que alguna sustancia o factor de los neumonococos lisos muertos
por el calor podía transferirse a la cepa rugosa, transformándola en
virulenta.
Entonces Griffith pensó que la solución a este rompecabezas era que en algún momento, y de alguna forma, las bacterias no infecciosas, rugosas, se habían transformado en bacterias virulentas. ¿Pero cómo? Pues incorporando el material genético de las bacterias muertas lisas, y adquiriendo la capacidad (perdida anteriormente) de producir la cubierta protectora, lo cual las había convertido en virulentas.
Con esta interrogante otros investigadores diseñaron nuevos experimentos y se llevaron a cabo una y otra vez ensayos que permitieran establecer cuál era esta misteriosa sustancia transformadora . No fue sino hasta 1944 que C.T. Avery, C.M. Mc Leod y MJ. McCarty publicaron sus resultados sobre la transformación bacteriana y demostraron que la sustancia transformadora era ADN. Estos estudios concluyeron que la sustancia transformadora no era más que el ADN de las bacterias lisas, que se había incorporado por el mecanismo de transformación bacteriana al ADN de las bacterias rugosas, haciéndolas virulentas. Es decir, por medio de la transformación bacteriana se pueden insertar fragmentos de ADN extraño en el cromosoma de otra bacteria, reemplazando al gene inactivo de esta última y recuperando su capacidad virulenta una vez más.
A pesar de que esto demostraba que el ADN era el material genético, pocos biólogos y genetistas estaban convencidos de haber encontrado, finalmente, la molécula de la herencia.
Vino entonces la prueba definitiva. Alfred Hershey y Margaret Chase en 1952 llevaron a cabo el experimento que no dejaría lugar a dudas de que el ADN era la molécula de la herencia, el material genético. Utilizaron un organismo novedoso, un virus capaz de destruir a las bacterias como Escherichia coli. Este bacteriófago o virus tiene un solo cromosoma de ADN dentro de una cápside de proteínas. Es muy bonito pues tiene forma de nave espacial y al momento de infectar lo hace como si se estuviera inyectando a la bacteria con un émbolo, quedándose la cápside en el exterior de la bacteria, mientras que el ADN es impulsado hacia adentro. El proceso de infección continúa con la incorporación del ADN viral en el cromosoma de la bacteria, apoderándose de su maquinaria genética para ordenar la fabricación de virus, los cuales llegan a romper la célula y son liberados hacia el exterior de ella, listos para infectar a otras bacterias.
Para demostrar que el ADN penetra en la bacteria, Hershey y Chase idearon hacer crecer al virus en un medio con fósforo y azufre radiactivos, es decir; marcándolo radiactivamente con estos dos compuestos. Como ya sabemos, las proteínas contienen azufre y no fósforo, por lo tanto, solamente la cápside del virus tenía azufre radiactivo. Y por el contrario, como el ADN contiene fósforo pero 110 azufre, el ADN viral estaba marcado con fósforo radiactivo, lo cual hacía muy distinguible su presencia dentro o fuera de la bacteria al momento de la infección.
Al infectar a las bacterias con el virus marcado se esperaría encontrar cuál
de los dos elementos, la proteína exterior o el ADN cromosomal,
penetraba en la bacteria y transformaba su aparato genético para producir más
virus. A este experimento se le denomina el experimento de la licuadora,
pues Hershey y Chase, una vez que hubieron infectado las bacterias, y sin dar
mayor tiempo para la reproducción interna de nuevos virus, metieron la mezcla
de virus y bacterias en una licuadora de cocina, para que, al agitarla, se desprendieran
las dos porciones del virus. Después de esto pudieron separar por centrifugación
los elementos y analizarlos. El resultado fue que la mayor parte del fósforo
radiactivo, es decir, el ADN, se encontró dentro de las bacterias
infectadas, y por el contrario, se encontró muy poco azufre radiactivo, es decir,
proteína marcada, dentro de las bacterias. Para Hershey y Chase, y para la comunidad
biológica, ésta era la prueba definitiva de que es el ADN es el
material genético (Figura 18).
Pero, ¿ cuál es su estructura?
Ya para 1950 se sabía que los ácidos ribonucleicos estaban formados por monómeros
de fosfatos, azúcares y bases nitrogenadas. Pero lo curioso es que Erwin Chargaff
demostró que, según el organismo, el ADN tenía diferentes proporciones
de las bases nitrogenadas. Al analizar las proporciones de estas bases en otros
organismos se dio cuenta de que siempre aparecían las mismas proporciones de
adenina y timinas y, por otro lado, de guaninas y citosinas. Pronto dedujo que
existía una regla general: las cantidades de A y T (adenina y timina) son iguales,
mientras que las de G y C (guanina y citosina) guardan también la misma proporción.
En el caso del humano y otros mamíferos las cantidades son aproximadamente:
21%C, 21%G, 29% A y 29%T. Independientemente del origen del ADN,
la regla de Chargaff se conserva: la mitad exacta de las bases nucleotídicas
son purinas (adenina y guanina), y la otra mitad son pirimidinas (timina y citosina).
Serían posteriormente Watson y Crick quienes interpretaran esta curiosidad
de Chargaff en términos del apareamiento de las bases en la molécula.
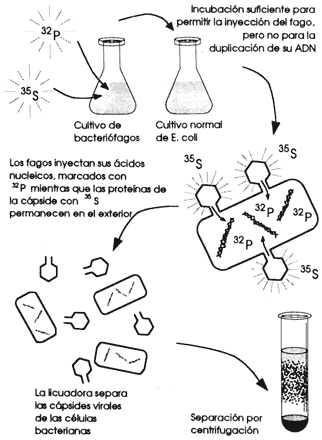
Figura 18. ¿Cuál es el material genético? Esta pregunta la cotestaron Harshey y Chase marcando fagos radiactivamente. La cúspide con azufre radiactivo y el ADN con fósforo radiactivo. Posteriormente infectaron una colonia de bacterias, pero sin dar tiempo para la duplicación del ADN. Separaron la cápside vacía del fago y encontraron que únicamente el fósforo radiactivo (32P) había penetrado en la célula.
Con la ayuda de la cristalografía de rayos X se pudo dilucidar la estructura del ADN en 1953. Esta técnica consiste en dirigir los rayos X a un cristal y observar cómo éstos son desviados (difractados) por la estructura molecular repetitiva dentro del cristal. Este patrón de difracción es posible verlo en una placa fotográfica, en donde encontraremos líneas y puntos distribuidos en forma radial, que permiten entender el acomodo de las moléculas de un cristal inorgánico. Gracias a que algunos compuestos orgánicos como el ADN, el ARN las proteínas pueden cristalizarse, fue exitosa la idea de estudiar el ADN con esta técnica, diseñada originalmente para cristales inorgánicos. Fue así como Maurice Wilkins y Rosalind Franklin obtuvieron fotografías del ADN y ciertas medidas intramoleculares (es decir las medidas que separaban a los constituyentes del ADN) cuyo significado desconocían, pero que aparecían con regularidad: 2.0 nanómetros (nm), 0.34 nm y 3.4 nm.
Fue así como James D. Watson y Francis Crick intervinieron en este problema sobre la dilucidación de la estructura de la doble hélice, el ADN. Partiendo de la regla de Chargaff sabían que el número de timinas era igual al de adeninas, y que el número de guaninas era igual al de citosinas. Pero, ¿cómo estaban dispuestas?
Sabían que tenía que haber una explicación para los números proporcionados
por Wilkins y Franklin. Hicieron varios modelos para probar diferentes disposiciones
de los elementos que constituyen el ADN, hasta. que uno de éstos
resultó apegarse con mayor exactitud a los datos de Chargaff y de Wilkins-Franklin.
El ADN parecía ser una doble cadena, enrollada sobre sí misma,
con un esqueleto de fosfato- azúcar (los pilares), con las bases nitrogenadas
(uniendo los pilares) orientadas hacia el interior. En este modelo los números
que hemos mencionado indicaban que el ancho total de la doble hélice era de
2.0 nm (nanómetros), el grosor de las bases nucleotídicas era de 0.34 nm., y
dado que la doble cadena gira sobre sí misma enrrollándose, la escalera de caracol
daría una vuelta completa cada 3.4 nm, esto es, cada 10 pares de bases. He aquí
el modelo de la doble hélice de Watson y Crick (Figura 19).

Por este gran descubrimiento recibieron tiempo después el Premio Nobel en fisiología y medicina en 1962. El descubrimiento de la estructura del ADN marcó una nueva etapa de la biología molecular.
Como ya hemos mencionado, el material genético tiene la capacidad de sintetizar otros compuestos (proteínas) y de duplicarse (hacer copias exactas de sí mismo).
El ADN tiene que duplicarse cuando la célula entra en división, para que cada célula hija tenga el mismo contenido de ADN, es decir, el mismo número de cromosomas.
El mismo modelo de Watson y Crick dio la respuesta a la duplicación. El ADN es como una escalera enrollada. Lo primero que tiene que ocurrir es que una enzima especial desenrolle la cadena hasta dejarla como si estuviéramos viendo una escalera sobre una pared. El segundo paso es separar a Watson y Crick, es decir, separar ambos lados de la escalera. Para esto existe una enzima especial, llamada ADN polimerasa, que se encarga de apartar a las dos cadenas; rompiendo los puentes de hidrógeno que antes las unían. Otra enzima se encarga de ir apareando las bases con sus contrapartes en cada cadena; en cuanto se van llenando los huecos la separación de las cadenas Watson y Crick continúa. Al final de este proceso tenemos dos dobles hélices donde antes sólo había una. A este tipo de duplicación se le conoce como semiconservadora pues ambas hélices nuevas tienen una de las cadenas originales (Figura 3).
¿ Cómo se procesa la información contenida en el ADN? El código genético
Como ya hemos mencionado la molécula de la herencia, el ADN, tiene
forma de una escalera de caracol, y cada uno de sus peldaños es un nucleótido.
Cada nucleótido está caracterizado por su base: adenina, guanina, timina y citosina.
La información genética es precisamente la secuencia de estas bases dentro de
un segmento determinado. Se dice entonces que la información está codificada
por su secuencia de bases. Cada tres bases forman un codón, que corresponde
a su vez a uno de los 20 aminoácidos existentes (Figura 20).
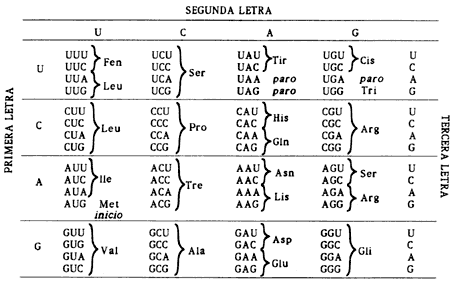
Figura 20. El código genético. En esta tabla podemos observar que existen
64 codones posibles. La columna de la izquierda marca la primera letra de los
codones. En la parte superior está la segunda letra y a la derecha la tercera
letra. Cada codón indica el aminoácido que produce. El código genético es redundante,
ya que existe más de un codón para cada aminoácido. UAA, UAG
y UGA representan señales de paro o término. AUG tiene
dos funciones: cooficia para metionina y también significa inicio. Todos los
ARM comienzan con el codón AUG, lo cual indica que
éste es el codón iniciador.
De esta manera, la información contenida en el ADN tiene que ser transcrita (o vuelta a escribir) a otra molécula intermedia, su similar, el ARN. Una cadena sencilla es transcrita a una molécula complementaria de ARN de acuerdo con las reglas establecidas por el modelo de Watson y Crick: una A se aparea con una U (recordemos que en el caso del ARN la timina es sustituida por el uracilo), y una G por una C. El resultado es la formación de una molécula conocida como ARN mensajero que a su vez será traducida (o decodificada) en una proteína. Cada codón (tres bases consecutivas) de ARN dirige la incorporación de un aminoácido particular para formar una proteína. De este modo tenemos que la formación de proteínas a partir de ADN es un proceso de dos pasos. Primero, a partir de una cadena de ADN se forma un ARN mensajero, es decir, el ADN es transcrito en ARN; segundo, este ARN mensajero es traducido para formar las proteínas.
Dicho en otras palabras, los genes codifican para proteínas; la expresión de
un gene se hace a través de una copia de sí mismo en el ARN, que
a su vez dirige la síntesis de las proteínas específicas. La vía molecular de
esta síntesis fue designada por F. Crick como el dogma central de la biología
molecular expresando los flujos de información de la siguiente manera:
| Transcripción. | Traducción. |
Replicación D
ADN" ARN"Proteínas
|
En algunos casos este diagrama puede invertirse, pero sólo en el paso que va de ADN a ARN, nunca de proteínas a ARN. Podemos obtener copias de ADN a partir de ARN si está presente una enzima llamada transcriptasa reversa, que como su nombre lo indica toma como molde una molécula de ARN y produce o sintetiza una molécula de ADN. Este descubrimiento ha sido muy exitoso pues ha demostrado la posibilidad de producir ADN nuclear a partir de las moléculas de ARN aisladas del citoplasma.
Si el ADN contiene cuatro tipos de bases que arreglados en codones
(series de tres) nos dan un total de 64 combinaciones, tenemos que traducir
estas 64 posibilidades en los 20 aminoácidos que se presentan en las proteínas.
De estos 64 codones se sabe que 61 codifican para aminoácidos, mientras que
los tres restantes indican el término de la síntesis. También podemos notar
en la figura 20 que varios tripletes pueden codificar para el mismo aminoácido,
de donde se dice que el código genético es redundante. A pesar de que
las investigaciones acerca del código genético fueron llevadas a cabo en la
región rII del bacteriófago T4, se han obtenido los resultados
que indican que este código es universal, es decir, se presenta en todos los
seres vivientes.
Ahora, es fácil suponer que si una proteína promedio está formada por 300 aminoácidos
(las hay más grandes, desde luego), y cada triplete o codón codifica para uno,
se requieren 900 pares de bases para formar la proteína. Se sabe que en algunos
organismos procariontes como Escherichia coli su cromosoma sencillo contiene
aproximadamente tres millones de pares de bases, suficientes para codificar
más o menos 3 000 proteínas. En las células eucariontes la cantidad de ADN
aumenta proporcionalmente con la complejidad. Por ejemplo, en células de mamíferos
existen aproximadamente de tres a cuatro billones de pares de bases formando
los cromosomas, cantidad suficiente para codificar más o menos tres millones
de proteínas, aunque se sabe que las proteínas presentes en este tipo de células
no son más de 100 000 y en algunos casos pueden ser hasta 30 000. Lo que sucede
es que existen secuencias que son meramente estructurales, y otras que están
repetidas hasta cientos de veces por genoma. Esto explicaría la cantidad tan
alta de ADN en relación con la cantidad de proteínas que pueden
procesarse. Otra explicación es que en el caso de los procariontes, la transcripción
y la traducción se llevan a cabo en el mismo lugar, mientras que en los eucariontes
estos dos procesos están separados tanto en tiempo como en lugar. El ADN
es transcrito dentro del núcleo en la molécula de ARN mensajero
precursor. Este precursor es procesado en el núcleo y reducido su tamaño para
formar un ARN mensajero maduro que sale a través de la membrana
celular hacia el citoplasma de la célula en donde es traducido a proteínas.
Esto quiere decir que no todo el ADN que se transcribe es traducido
a proteínas. A este problema volveremos más adelante, por ahora volvamos a la
pregunta de cómo tal cantidad de ADN puede estar contenida dentro
del núcleo de las células eucariontes.
Pero, ¿cómo se forman los cromosomas?
Como ya hemos visto, en las células eucariontes no todo el ADN se encuentra en el núcleo, también encontramos ADN en las mitocondrias (organelos donde se lleva a cabo la respiración) y en los cloroplastos, en el caso de las células vegetales (en ellos se lleva a cabo la fotosíntesis). Sin embargo, podemos decir que casi todo el ADN se encuentra en el núcleo, que de manera ordinaria está dividido en dos o más cromosomas. El número de éstos es característico de cada especie: el hombre tiene 23 pares de cromosomas, el maíz 10, la mosca de la fruta 4, etc. La cantidad de ADN presente en cada núcleo celular es tan grande que, como veremos más adelante, necesita enrollarse y doblarse para formar los cromosomas.
En las células eucariontes el ADN es el principal componente del
núcleo, pero no se encuentra suelto sino formando parte de la cromatina.
En 1879 Flemming usó este término de cromatina (del griego chroma, color)
para designar a la sustancia que toma tonalidades intensas con colorantes básicos
en el núcleo de las células debido a la presencia de una sustancia llamada por
él nucleína, compuesto fosforado que se había aislado en 1871 de células de
pus.
Ya desde 1876 Balbiani había observado que antes de la división celular en el núcleo se formaban pequeñas estructuras cilíndricas, y más tarde en 1888 Waldeyer denominó a estas estructuras cromosomas, haciendo énfasis en la continuidad entre la cromatina descrita por Flemming y las estructuras cilíndricas explicadas por Balbiani.
Gracias al desarrollo de ciertas técnicas citológicas se pudo aislar la cromatina del núcleo que es una masa gelatinosa compuesta por ADN, ARN, proteínas básicas —histonas— y proteínas no histónicas o ácidas. La cantidad de ARN y de proteínas no histónicas es variable de célula a célula, pero la cantidad de histonas nos revela una relación de uno a uno con el ADN. Estas proteínas son de cinco clases y su papel es proporcionar una estructura estable al ADN. Estas histonas se unen con fuerza al ADN para formar una estructura llamada nucleosoma.
Cuatro de las cinco histonas forman octámeros, alrededor de los cuales se enrolla
el filamento de ADN que da dos vueltas de 140 pares de bases. La
quinta histona se une al puente de ADN que une los nucleosomas,
los cuales a su vez se pliegan para formar una fibra gruesa en la cual hay seis
nucleosomas por cada vuelta de la doble hélice (Figura 21).
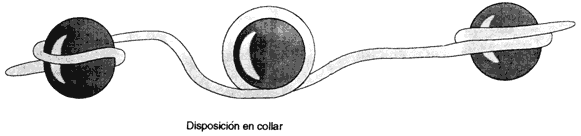
Figura 21. Relación de la fibra de ADN con la histona del nucleosoma. Las histonas están representadas por las pelotas y el ADN por el tubo. El ADN se enrolla por fuera de las proteínas (histonas) dando configuración en collar.
Esto quiere decir que en el núcleo el ADN está formando la cromatina
en asociación con ARN y proteínas; cuando se va a iniciar la división
celular los nucleosomas se superempaquetan, formando los cromosomas. (Figura
22). Es por esta razón que una fibra tan larga como el ADN ocupa
un espacio muy pequeño dentro del núcleo de las células, haciendo más fácil
la división celular. Esta organización del ADN en los cromosomas
explica por qué todos los datos requeridos para la formación de una planta,
un animal o una persona se puede guardar dentro del núcleo de las células eucariontes.
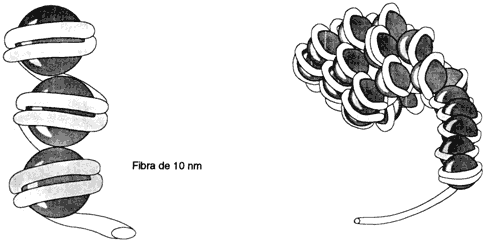
Figura 22. Enrollamiento de la fibra 10 nm para constituir la de 20-30 nm.
La historia de las ciencias demuestra que el desarrollo de una ciencia no sólo depende de la genialidad de los investigadores o científicos, sino de la existencia de un horizonte teórico común que permita el entendimiento y asimilación de las ideas; y también enseña que, algunas veces, muchos de los descubrimientos científicos quedan fuera del entendimiento común. En la historia de la genética tal es el caso de Mendel, cuyas leyes quedaron en el olvido o desconocimiento durante cerca de 40 años, en espera de un ambiente científico que permitiese su asimilación a principios del siglo XX. A partir de su reconocimiento universal, el mendelismo constituyó la piedra angular de la genética moderna. Otro caso similar es el de Bárbara McClintock, quien en 1948 descubrió un fenómeno genético hasta entonces inimaginado: la transposición o movilidad de los genes dentro de los cromosomas. A pesar de que sus estudios pasaron inadvertidos para la comunidad científica del momento durante casi 30 años (en la segunda mitad del presente siglo, florecimiento de la biología molecular), la historia le hizo justicia cuando se le otorgó el Premio Nobel en fisiología y medicina en el año de 1983 por sus experimentos de los genes saltarines con la planta del maíz.
¿ Cómo fueron descubierto los genes saltarines ?
Bárbara McClintock empezó su trabajo de genética con la planta del maíz. Encontró que las semillas de las mazorcas mostraban coloraciones diversas, a veces otras mostraban pequeños parches verdes, blancos o amarillos pálidos. De esta observación, McClintock dedujo que cada parche indicaba la presencia de una familia de células que en algún momento del desarrollo había sufrido algún cambio o mutación. Dependiendo del momento del cambio (mutación), el parche podría adoptar diferentes coloraciones y tamaños. Si era temprano los fragmentos serían grandes, mientras que si era tardío el parche sería pequeño. De igual forma, la cantidad de parches presentes en una semilla indicaría que el cambio o mutación había ocurrido múltiples veces. Así fue como B. McClintock decidió seguir la historia celular y averiguar qué era lo que había ocurrido.
Haciendo análisis citológicos al microscopio, McClintock analizó el comportamiento de los cromosomas durante la división celular. En particular, el par número 9 (la planta del maíz tiene pares de cromosomas) mostraba ciertos rompimientos que ocurrían con gran regularidad y en lugares específicos. Durante sus observaciones notó también que una vez ocurrido un rompimiento, éste permanecía en las generaciones celulares siguientes. McClintock llegó a la conclusión de que no era más que un tipo de rompimiento controlado en el cromosoma, y que cada tipo de rompimiento correspondía a una clase de parche en la semilla, el cual determinaba también su coloración.
Estudiando así la herencia del color y la distribución de estos parches de la planta del maíz que había presentado rompimientos cromosómicos repetidos, B. McClintock encontró que la actividad de genes particulares se había modificado gracias a estos rompimientos. Como estos genes estaban asociados a la coloración de los granos de las mazorcas, algunas de éstas aparecían moteadas y otras sin coloración. Esta actividad, concluyó B. McClintock, se debía a la acción de unidades génicas, a las que llamó elementos controladores, que aparentemente podían moverse de lugar dentro del cromosoma originando que se modificara la actividad de otros genes, junto a los cuales estos elementos se insertaban. Sus análisis microscópicos demostraron la existencia de estos elementos controladores y confirmaron que éstos servían como sitios específicos de rompimiento y salida de segmentos de ADN, causando cambios pequeños y grandes.
En 1948 B. McClintock publicó sus resultados y su hipótesis: existen elementos genéticos capaces de produrcir los rompimientos en los cromosomas y alterar los patrones de desarrollo de las células. Estos elementos genéticos no son más que segmentos de ADN (genes) capaces de moverse o saltar dentro de los cromosomas, produciendo un cambio o mutación en el lugar donde se inserten. A este fenomeno McClintock lo denominó transposición.
La transposición es la movilidad que poseen ciertos genes dentro del genoma celular de un organismo. En la actualidad se conocen y se han clasificado varios segmentos de ADN con estas características, que son: secuencias de inserción, transposones, y ADN extracromosomal, como los virus y los plásmidos.
Las secuencias de inserción (SI) son los elementos móviles más pequeños, pues consisten de unas cuantas bases de nucleótidos. Los transposones son elementos más grandes que incluyen SI en sus extremos con genes intermedios; estos extremos repetidos permiten a toda esta unidad salirse de un sitio e insertarse en otro. Los plásmidos son segmentos de ADN extracromosomal que tienen la capacidad de insertarse en el cromosoma bacteriano, o pasar de bacteria en bacteria. Los virus pueden funcionar como elementos móviles, ya que cuando infectan una bacteria o una célula eucarionte se insertan y separan de la misma forma que los transposones.
Aunque la estructura de estos elementos sea diferente, sobre todo por su tamaño, todos comparten características importantes: pueden insertarse en los cromosomas gracias a la presencia de terminales definidas por secuencias específicas que hacen que reconozcan los lugares donde han de insertarse y también son capaces de duplicarse independientemente de la replicación de todo el genoma durante la duplicación celular.
Estos elementos causan mutaciones o alteraciones, así como reacomodos de los genes que ya existen, provocando que la expresión de éstos sea diferente. También pueden apagar o encender los genes junto a los cuales se inserten, así como viajar de bacteria en bacteria confiriéndole, por ejemplo, resistencia a ciertos antibióticos.
Posteriores estudios han comprobado la existencia de estos elementos en otros organismos. Además del maíz, se les ha encontrado en la mosca de la fruta Drosophila melanogaster, en hongos, en bacterias, y en el hombre y se cree que ésta sea una característica universal de todos los organismos vivientes.
Una de las más importantes consecuencias de este fenómeno de la transposición está relacionada con la medicina: ¿cómo es que las bacterias han adquirido la resistencia a ciertos antibióticos? Debido a la gran administración de antibióticos en la medicina humana y en veterinaria, la transposición ha adquirido dimensiones mayores. Antes de este descubrimiento se sabía que la resistencia a diversos antibióticos podía ser transmitida de bacteria a bacteria a través de los plásmidos —segmentos de ADN que no forman parte del cromosoma bacteriano—, pero no se entendía con claridad cómo es que estos genes se acumulaban en ellos. La explicación actual radica en que los elementos que otorgan la resistencia a los antibióticos en los plásmidos no son más que colecciones de transposones que contienen genes que codifican para la resistencia, a uno o a varios antibióticos.
P. Sharp descubrió que la estructura de ciertos plásmidos bacterianos es modular. Esto es, que ciertas partes del plásmido son muy parecidas entre sí, mientras que otras no muestran parecido alguno. Esto se explica debido a la abundante replicación de secuencias dentro del plásmido. Se han encontrado en la naturaleza transposones idénticos en ciertas especies bacterianas. Es así como distintas especies de bacterias han adquirido en el curso de la evolución la capacidad para resistir a los antibióticos. Gracias al descubrimiento de la transposición muchos de estos rompecabezas se han resuelto, y esperemos que crezca su repercusión en la medicina.
LA ESTRUCTURA MOLECULAR DEL GENE
Cómo varía el material genético: la mutación
Los organismos actuales difieren no sólo en la suma total de su material genético, número de cromosomas, etc., sino también en su constitución estructural, fisiológica o de comportamiento. Denominamos genotipo a la constitución genética de un organismo, y fenotipo a la suma de las característica de una célula o de un organismo que resulta de la interacción del genotipo con el medio ambiente.
Si suponemos que todos los organismos vivos se derivan de un organismo ancestral con un genotipo determinado, entonces sería lógico pensar que el genotipo de este organismo sufrió cambios sucesivos en el curso de la evolución hasta producir la multitud de genotipos diferentes que ahora conocemos. A estos cambios en el genotipo se les llama mutaciones, y al producto se le denomina mutante.
Todos aquellos cambios genéticos que no se deban a la recombinación, son mutaciones por definición. Este término incluye una multiplicidad de fenómenos como cambios físicos en los genes, cambios cromosómicos estructurales y cambios en el número de éstos.
Podemos dividir a las mutaciones en grandes (macromutaciones) y pequeñas (micromutaciones). Empezaremos por analizar las pequeñas.
Estas mutaciones son causadas por sustitución, adición o eliminación de nucleótidos
dentro de una sección o gene de ADN o ARN. Este cambio
puede producir algún cambio fenotípico visible. Es decir, algunas de estas mutaciones
puntuales pueden ser silenciosas, no observables por métodos convencionales,
pero detectables con análisis moleculares.
El modelo de Watson y Crick sugirió que si un par de nucleótidos era sustituido o sustraído causaría una mutación puntual. Más tarde, Freese en 1959 mapeó las mutaciones producidas en el fago T4 por una base análoga a las cuatro que ya conocemos, la 5-bromouracil y observó que los agentes usados requerían que el ADN se replicase para producir una mutación. Es decir, es durante el proceso de replicación de la molécula del ADN que se presentan las mutaciones puntuales. A partir de estas observaciones, Freese propuso dos diferentes clases de mutaciones puntuales: las transiciones y las transversiones.
Las transiciones son el reemplazo de una purina por otra purina o de una pirimidina
por otra pirimidina, causando una mutación en la cual la proteína tiene un aminoácido
sustituido por otro. Si esta mutación se lleva a cabo durante la replicación
del ADN, la mutación se fijará en la siguiente replicación (Figura
23(a)).
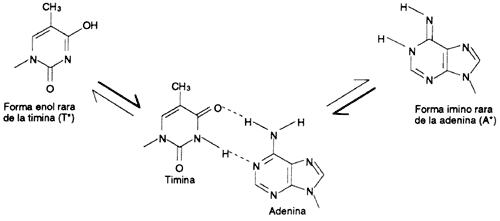
Figura 23(a). Transiciones y transversiones.
Las transversiones son el remplazo de una purina por una pirimidina, y viceversa,
causadas por ciertos compuestos como las acridinas. Estas mutaciones producen
proteínas que no son similares a la original (Figura 23(b)).
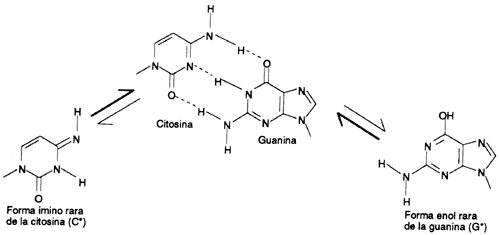
Figura 23(b). Transiciones y transversiones.
Mutaciones estructurales en los cromosomas
Las mutaciones estructurales en los cromosomas pueden ser de varios tipos:
1) Por pérdida o ganancia de alguno de los genes en un cromosoma. Si un cromosoma normal contiene los genes ABCDEFG, el cromosoma deficiente contendrá sólo los genes ABCDFG; si el cromosoma normal contiene los genes ABCDEFG, el cromosoma duplicado contendrá los genes AABBCDEFG.
2) Por cambios debidos a una alteración en el agrupamiento de los genes. Estos cambios son causados: i) por desplazamiento o translocaciones que indican que una parte de un cromosoma se adhiera a otro cromosoma diferente, o que haya intercambio de secciones entre dos cromosomas; ii) por inversiones que indican que en el mismo cromosoma alguna sección gire sobre su eje 180°, originando una inversión de la secuencia de genes; por ejemplo, si en un cromosoma los genes están en el siguiente orden: ABCDEFG, una inversión daría como resultado el siguiente orden: AFEDCBG. iii) por transposición lo cual indica que un bloque de genes se desplaza a una nueva posición dentro de un cromosoma, por ejemplo ABCDEFG muta por transposición a ADEFBCG.
Cambios numéricos en los cromosomas.
Se puede alterar el número de cromosomas por:
1) aneuploidia, que es la falta de uno o más cromosomas en el juego normal (por ejemplo el síndrome de Turner; que indica la falta de un cromosoma del par sexual en las mujeres), o un exceso de ellos (como el síndrome de Down, que indica la presencia de tres cromosomas en el par 21 del hombre); y
2) poliploidia, que produce organismos con dos o más juegos de cromosomas. Este fenómeno es frecuente en plantas y se sabe que gracias a este evento se han generado especies nuevas.
Qué efectos producen estas mutaciones
Si los genes codifican para alguna proteína, su cambio o mutación afectará
la producción de esta última. Esto fue demostrado por Beadle y Tatum en 1948
cuando propusieron una cadena de producción de la proteína en la cual si un
paso era afectado, el resultado era la falta de formación de la proteína final.
Este es el caso de las enfermedades metabólicas, las cuales indican un error
genético heredable. En el hombre se conocen varias de estas enfermedades que
son producidas por mutaciones específicas en el ADN, que alteran
la producción normal de las proteínas. Por ejemplo, la fenilcetonuria
es una enfermedad que causa retraso mental en el hombre. En 1930 se descubrió
que en la orina de los pacientes que sufrían esta enfermedad estaba presente
una sustancia llamada ácido fenilpirúvico. En este caso, la fenilalanina, componente
normal en la dieta, no puede ser degradado a tirosina debido a la ausencia de
la enzima que lleva a cabo este paso metabólico, entonces la fenilalanina es
transformada por una vía alterna en dosis peligrosas de ácidos fenilpirúvico,
produciendo el retraso mental de los pacientes. Ahora podemos decir que esta
enfermedad es producto de una mutación que inhibe la producción de la enzima
presente en el metabolismo normal. Existen otras enfermedades metabólicas cuyos
rasgos característicos son la ausencia de enzimas por la mutación de un gene
particular (Figura 24).
| |
|
| Enfermedad |
Enzima o proteína afectada |
| |
|
| Acatalasemia | Catalasa |
| Afibrinogenemia | Fibrinógeno |
| Agammaglobulinemia | Gamaglobulina |
| Albinismo | Tirosinasa |
| Alcaptonuria | Oxidasa del ácido homogentísico |
| Analbuminemia | Albúmina sérica |
| Galactosemia | Trasferasa de la uridil-galactosa 1-fosfato |
| Enfermedades con acumulación de glucógeno: | |
| Tipo I (Von Gierke's | Glucosa 6- fosfatasa |
| Tipo III | Amilo-1, 6-glucosidasa |
| Tipo IV | Amilo-(1,4 - 1,6) transglucosidasa |
| Tipo V (Mc Ardle's) | Fosforilasa del músculo |
| Tipo VI (Hers') | Fosforilasa del hígado |
| Bocio familiar | Deshalogenasa de la yodotirosina |
| Hemoglobinopatías | Hemoglobinas |
| Hemofilia A | Factor antihemofílico A |
| Hemofilia B | Factor antihemofílico B |
| Histidinemia | Histidinasa |
| Hiperbilirrubinemia (enfermedad de Gilbert) | Transferasa de uridino-disfosfato glucoronato |
| Hipofosfatemia | Fosfatasa alcalina |
| Sindrome de Lesh-Nyhan | Trasferasa de fosforribosil-hipoxantina-guanina |
| Metahemoglobinemia | Metahemoglobina-reductasa |
| Fenilcetonuria | Fenilalanina-hidroxilasa |
| Enfermedad de Wilson | Ceruloplasmina |
| Xantinuria | Xantinooxidasa |
| Xerodermia pigmentaria | Enzimas que reparan el ADN |
| |
|
Figura 24. Enfermedades hereditarias en el hombre en las que falta o se
ha modificado una proteína.
Como ya hemos mencionado, la biología molecular es una rama derivada de la genética mendeliana cuyo objetivo es el análisis estructural, bioquímico y morfológico del material genético. La introducción de microorganismos como las bacterias y los virus tuvo consecuencias importantes en el desarrollo de esta rama ya que permitió trabajar muchas generaciones en poco tiempo, con poco material genético (procariontes) y con una capacidad de manipulación que era impensable tanto para la época en que se hicieron experimentos con la mosca de la fruta Drosophila melanogaster como para los años en los que Mendel trabajó con plantas.
Una de las características de la genética bacteriana es que los mutantes que se producen pueden estudiarse desde el punto de vista bioquímico. Esto es, pueden analizarse los productos que resultan de la alteración del gene bacteriano. A los mutantes que han perdido su capacidad para crecer en cierto tipo de medios se les llama auxótrofos. Estos mutantes son muy útiles, ya que podemos inferir la composición genética de una bacteria a partir de su crecimiento en cierto medio dentro de una caja de Petri. Dicho en otras palabras, si sabemos que cierta bacteria, digamos A, crece normalmente en un medio con la sustancia X, y producimos una mutación que impida que esta bacteria A crezca con la sustancia X, entonces podremos analizar bioquímicamente su comportamiento. Podemos decir que la mutación afectó una sección del cromosoma bacteriano (gene) que ha bloqueado la formación de la enzima que digiere a la sustancia X por medio de la cual la bacteria obtiene sus recursos alimenticios. Si infectamos una cepa A auxótrofa para la sustancia X, con un virus conocido, el genoma viral por traducción se incorporará al cromosoma bacteriano. Si el fragmento incorporado del virus lleva el gene normal del mutante auxótrofo, es decir, su capacidad para digerir a X, la cepa restablecerá su función original. Si se lleva a cabo la transformación de las bacterias podremos separar a los grupos dentro de la caja de Petri fácilmente: aquellos que han incorporado el genoma viral crecerán formando placas definidas en la caja, aquellas que no lo hayan hecho simplemente morirán ya que, recordemos, son auxótrofas para la sustancia X. ¿Que significado tiene esto? Estos experimentos sugieren que un gene es una región extendida dentro del cromosoma y que los cambios que ahí ocurren pueden dar lugar a diferentes mutantes, dependiendo de la posición de la mutación o inserción de nuevas secuencias de ADN. También indica que las regiones dentro de esta sección o gene son separables y recombinables con regiones homólogas de un gene de otro cromosoma.
Con estos estudios Seymour Benzer pudo caracterizar, según las propiedades moleculares, el tamaño de las unidades de función, de mutación y de recombinación.
S. Benzer en 1957 definió el gene de un modo novedoso que incluye una división tripartita del gene clásico (mendeliano) basada en la estructura fina de la mutación, la función y la recombinación. Estos análisis de Benzer demostraron que dentro de los genes había secciones que podían ser intercambiables con sus homólogos del otro cromosoma. La unidad de recombinación fue definida como el elemento más pequeño dentro de un gene que es intercambiable por recombinación genética. A este elemento se le llamó recón. La unidad de mutación, el mutón, se definió como el elemento más pequeño que, alterado, da lugar a una forma mutante del organismo. La unidad de función, definida genéticamente, fue definida como el segmento del mapa que corresponde a una función unitaria llamándosele cistrón.
La definición del gene como unidad de función y de mutación se tomó obsoleta a partir de estos estudios sobre la estructura fina del gene. En 1957 Benzer simplificó la definición del gene clásico, dividiéndola en cistrón, mutón y recón. Al gene que produce una proteína específica se le denominó cistrón, al segmento más pequeño que sufre alteraciones, mutón, y al gene definido como el segmento capaz de recombinarse, recón. De estos términos, el más ampliamente usado, especialmente en sistemas microbianos como sinónimo de gene, pero con una clara connotación que hace referencia a la estructura molecular encontrada por Benzer, es el de cistrón.
Desde que los trabajos de Mendel fueron reconocidos de manera universal a principios de este siglo, hemos visto que los conceptos originales de carácter unitario, factor o gene, se han modificado debido al avance tanto teórico como experimental de la genética, que ha permitido mirar más de cerca a las unidades de la herencia. Durante estos primeros años de desarrollo se creía que los genes eran unidades discretas que producían un carácter particular a través de la síntesis de una proteína. Posteriormente, y gracias al modelo de Watson y Crick se supo que el gene estaba constituido de ADN, formando una "doble hélice". Se entendieron, en consecuencia, la replicación o duplicación, la mutación o variación del material genético, y la síntesis de proteínas. Sin embargo, investigaciones más recientes han demostrado que los genes no son continuos, sino que si los miramos más de cerca, están divididos o partidos en trozos o secciones.
Este descubrimiento indicó que los genes están fragmentados en una serie de
regiones alternantes que codifican para partes de las proteínas (exones) y regiones
intercaladas (intrones) cuya información no indica la creación de ningún compuesto.
Los exones y los intrones forman una unidad que es transcrita como una sola
molécula de ARN. Recordemos que del ADN se forma una
molécula de ARN mensajero precursor dentro del núcleo, que es procesado
para formar una molécula de ARN mensajero maduro que sale del núcleo
para ser traducido a proteínas. Este proceso de separación de la transcripción
y la traducción puede explicar las discrepancias existentes entre la cantidad
de ADN que tiene una célula y la cantidad de proteínas que sintetiza.
Pero la pregunta obligada es: ¿qué segmentos son cortados de la molécula de
ARN mensajero antes de salir del núcleo?
Uno de los experimentos que llevó al descubrimiento de los genes partidos fue el de P. Chambon y sus colaboradores en 1975, quienes trabajaban con la proteína ovoalbúmina. Esta proteína tiene una cadena de 386 aminoácidos sintetizada en células glandulares especializadas del oviducto de las gallinas ponedoras. La diferenciación de estas células y la expresión del gene de la ovoalbúmina están controladas por las hormonas sexuales de las hembras. Si las hormonas no están presentes, el gene no se expresa y no se forma el ARN mensajero, y por lo tanto la ovoalbúmina no es sintetizada. Para entender esto, P. Chambon aisló el gene de la ovoalbúmina de células glandulares y también de otras células en donde este gene no es expresado (por ejemplo, en eritrocitos) para analizar la estructura molecular del gene en dos ambientes distintos.
El primer resultado que obtuvo fue que ambos genes eran idénticos. Entonces decidió comparar el gene de las células glandulares con el ARN mensajero aislado del citoplasma. El resultado fue que el ARN mensajero era de dimensiones menores que el gene original. Esto hizo suponer que el gene estaba partido en secuencias que eran codificadoras y en otras que no lo eran, a las primeras se les llamó exones, mientras que a las segundas, intrones. Análisis más finos de la estructura molecular del gene de la ovoalbúmina demostraron que éste estaba formado por ocho exones, entre los cuales se encontraron intrones que no tenían su contraparte en el ARN mensajero. La secuencia estudiada confirmó que el orden de los exones correspondía al orden de sus transcriptos en el ARN mensajero, indicando una colinearidad entre el ADN y el ARN. También pudieron medir el largo total de ambas moléculas. El tamaño total del gene era de 7 700 pares de bases, mientras que el mensajero contenía sólo 1 872.
El descubrimiento de los genes partidos explica por qué no todo el ADN codifica para proteínas. A este descubrimiento han seguido otros tantos que han demostrado una estructura similar para otros genes: el gene de la beta-globina (componente de la hemoglobina) de ratón y de conejo, por ejemplo. También se ha comprobado que están presentes en genes de mamíferos, aves y anfibios, algunos insectos, hongos, y se cree que sea una estructura universal de los genes de los organismos eucariontes.
Regulación y control genético: el modelo del operón
Hasta ahora nos hemos dedicado a comprender cómo está estructurado el ADN en los organismos, pero la siguiente pregunta por contestar es qué y cómo hace lo que tiene que hacer. Su tarea es, básicamente, construir los organismos. Siempre ha sido difícil imaginarse cómo de una molécula de ADN se obtienen bacterias, perros o animales tan espeluznantes como las víboras, los murciélagos, etc. Para darnos una idea del campo de la biología que estudia este desarrollo revisemos de nuevo algunos conceptos importantes.
Ya hemos hablado de los conceptos genotipo y fenotipo, el primero, designa el acervo genético de un individuo o una especie, y el segundo su apariencia. Sin embargo, poco hemos hablado de cómo el genotipo se expresa en el fenotipo, es decir, cómo el genotipo produce las características morfológicas, fisiológicas y de comportamiento que caracterizan a cada especie biológica.
La vía molecular de la síntesis de proteínas llamada por Crick el dogma
central de la biología molecular expresa los flujos de información de la
siguiente manera:
| Transcripción. | Traducción. |
Replicación D
ADN" ARN"Proteínas
|
Este aparato genético está sujeto a regulación. No todos los genes están actuando en todo momento, por lo cual la célula debe regular activando o desactivando aquellos genes cuyos productos le sean necesarios para responder satisfactoriamente al medio ambiente. (Si todos los genes de las células actuaran en todo momento, habría células semejantes y con las mismas funciones, cosa que evidentemente no ocurre; para que ocurra la diferenciación deben activarse algunos genes, mientras que otros permanecen inactivos.) Además, esta regulación está íntimamente relacionada con el crecimiento y la diferenciación celular.
Empezaremos hablando de la regulación en procariontes y posteriormente analizaremos la regulación en eucariontes.
En 1961 dos microbiólogos franceses, Francois Jacob y Jacques Monod, descubrieron un mecanismo de regulación y control de síntesis de proteínas en bacterias al que dieron el nombre de operón.
Notaron que la bacteria Escherichia coli sintetizaba ciertas enzimas de manera constante, mientras que otras se sintetizaban en el momento en que eran requeridas. Es decir, si la bacteria era colocada en un medio que contenía lactosa (azúcar de la leche), ésta empezaba a producir las enzimas que la digieren en galactosa y glucosa; por el contrario, si el medio carecía de lactosa, la bacteria no la producía pues era inútil su producción.
Jacob y Monod estudiaron la producción de tres enzimas que intervienen en la
digestión de la lactosa: la beta-galactosidasa, la galactosa permeasa y la tiogalactósido
transacetilasa. Por razones de nomenclatura decidieron llamarlas enzimas z,
y y a, respectivamente. De igual forma designaron a los genes que
las producían como los genes estructurales responsables de su síntesis (un gene
estructural es aquel que codifica, es decir que tiene la información necesaria
para la síntesis de una proteína o parte de ella) (Figura 25 (a)).
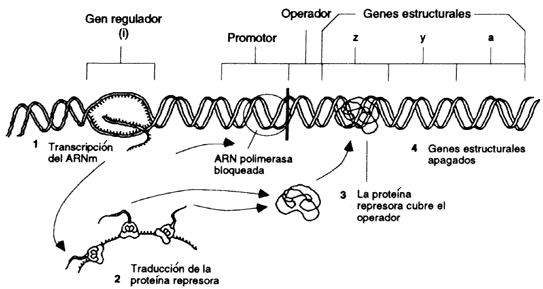
Figura 25. Modelo del operón de Jacob y Monod. En este modelo se ilustra la producción de las tres enzimas responsables del metabolismo de las lactosas que está bajo el control del operón lac. Este sistema está integrado por 3 partes: el gene regulador, la región promotora/reguladora y los genes estructurales z, y y a. En (a) los genes estructurales están reprimidos por la integración del gene regulador y el operador. 1) el gene regulador se transcribe en ARN mensajero, 2) es traducido en una proteína represora que, 3) se pega al operador y lo bloquea y 4) se inhibe la transcripción del ARNm en los genes estructurales.
Jacob y Monod descubrieron que los genes estructurales del operón de la lactosa
z, y y a, estaban alineados en el cromosoma, mientras
que un cuarto gene se localizaba a cierta distancia, al que llamaron gene regulador.
De estos cuatro genes, los tres estructurales se transcriben al mismo tiempo
en ARN, mientras que el cuarto gene, el represor, transcribe un
ARN mensajero que codifica para una proteína represora que se une
a otra región muy especial de ADN presente en el operón de la lactosa.
Esta región es la del operador. Si la enzima que sintetiza el gene regulador
se pega al operador, inhibe su acción y podemos decir que el sistema está apagado.
Si, por el contrario, esta enzima represora se pega con el azúcar de la lactosa
presente en el medio, se despegará del operador y será entonces cuando empiecen
a funcionar los genes estructurales, sintetizando las enzimas que desdoblan
eficientemente a la lactosa. Este sistema del operón de la lactosa que regula
la producción de enzimas se autorregula en el momento que las enzimas producidas
por los genes estructurales han digerido a la lactosa en galactosa y glucosa,
lo que disminuye la concentración de lactosa; y por lo tanto, la molécula represora,
al no encontrar lactosas libres para pegarse, se vuelve a unir al operador,
apagándolo. Este sistema, como vemos, es negativo y el inductor, el elemento
que provoca la reacción en cadena es, en este caso, la lactosa (Figura 25(b)).
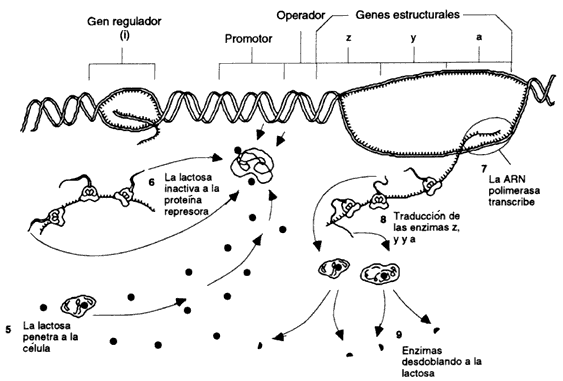
Figura 25(b). Observamos que cuando entra lactosa el medio 5), actúa como
inductor despegando la proteína represora 6), liberándose al operador, y la
ARN polimerasa inicia la transcripción. (8) el ARN
se traduce en tres enzimas del metabolismo de lactosa. Cuando la lactosa se
consume 9) la proteína represora puede unirse de nuevo al operador y cerrar
el sistema del operón lac.
Jacob y Monod no sólo estudiaron la estructura del operón de la lactosa, sino
también el operón del triptófano (Figura 26 (a)). El triptófano es un aminoácido
que la bacteria necesita constantemente en la síntesis de las proteínas necesarias
para su supervivencia. Jacob y Monod se dieron cuenta de que el operón del triptófano
(el cual controla las enzimas encargadas de la síntesis del triptófano) trabaja
constantemente, a menos que exista suficiente triptófano en el medio.
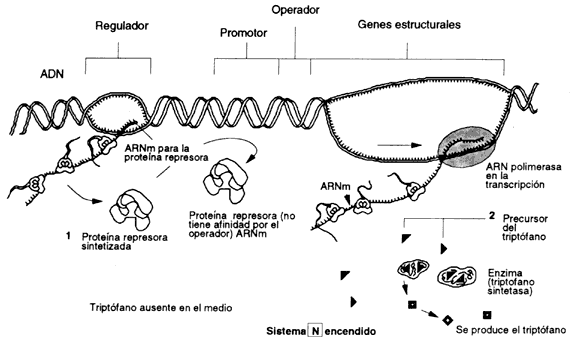
Figura 26(a). Operón del triptófano. Este sistema funciona de forma opuesta al operón de la lactosa. Aquí el operón se encuentra encendido ya que, aunque se sintetiza la enzima triptófano sintetasa, la cual es esencial para la producción del aminoácido triptófano 1), ésta no se une al gene operador.
Este funciona como el descrito para la lactosa, con la salvedad de que en el
primer caso la lactosa actuaba como inductora pegándose al operador; en el segundo
caso, el triptófano actúa como represor al unirse con una proteína represora,
formando un complejo triptófanorepresor que se pega al operador y que impide
la acción de los genes estructurales, por lo tanto no se sintetizan las proteínas
adecuadas. Como vemos este sistema también es de control negativo (Figura 26
(b)).
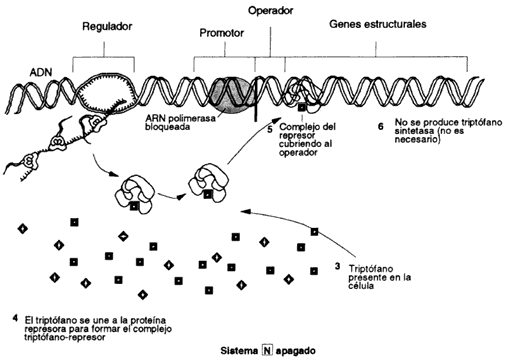
Figura 26(b). Observamos que si se añade al sistema el aminoácido triptófano 3), éste se une inmediatamente a la proteína represora 4) formando el complejo triptófano-represor, que a su vez se pega al operador y bloquea la acción de la ARN polimerasa en el promotor. En 5) se detiene a los genes estructurales 6) que producen la triptófano sintetasa.
A partir de la publicación en los años sesenta de estos hallazgos de Jacob y Monod se ha ido descubriendo una serie mayor de operones en diferentes organismos, pero todavía no hay pruebas concluyentes acerca de si su organización es universal para todos los organismos.
En los eucariontes la regulación genética es completamente distinta. Una de estas diferencias es su complejidad. Uno de los sistemas de regulación estudiados es el caso de la producción de hormonas esteroides en las gallinas. Los oviductos de las gallinas jóvenes responden fácilmente a una hormona femenina llamada estrógeno (que es un esteroide). En presencia de estrógeno, el oviducto empieza a sintetizar albúmina y demás proteínas de la clara del huevo. Se ha marcado radiactivamente al estrógeno que penetra en las células y se ha encontrado que se une a un receptor específico que es más que una proteína citoplasmática. Este primer complejo esteroide receptor entra en el núcleo de la célula y se adhiere a una proteína no histónica del cromosoma, iniciando así la transcripción al ejercer un control positivo.
Aunque nada está dicho en forma definitiva, estos estudios prometen dilucidar las diversas formas de control, por ejemplo de los anticuerpos, las hormonas, etc. Parece que tendrán que pasar algunos años y proponerse nuevas técnicas antes de que este tipo de problemas encuentren las soluciones adecuadas.
Como hemos visto, el desarrollo de la biología molecular ha sido vertiginoso desde su nacimiento en los años cincuenta hasta ahora. También hemos visto cómo este desarrollo ha repercutido de manera sobresaliente en otras ramas de la misma biología y la medicina. La biología molecular se ha convertido hoy en día en un arma poderosa para entender, asimilar y manipular el genoma de los organismos vivos. Esta manipulación, muy especial, ha sido posible gracias al desarrollo de técnicas nuevas cuyo poder de resolución ha permitido meternos dentro del ADN y no sólo explicarlo y describirlo, sino también jugar con él de manera satisfactoria.
Si pensamos en los primeros genetistas como Mendel, Morgan e incluso Muller (quien fuera el primero en producir mutaciones con radiación) resulta hasta inocente su forma de concebir la genética. Caracteres diferenciantes, proponía Mendel; ¿cómo se heredan las mutaciones? preguntaría Morgan, y ¿cómo producirlas a nuestro antojo? pensaría Muller. Ahora estas preguntas resultan como un juego de niños comparadas con las preguntas que la biología molecular intenta resolver. Sin embargo, hay que hacerles justicia: sin la solidez de sus hipótesis ni la claridad de sus experimentos, la ciencia de la genética nunca hubiera podido establecer un programa de investigación que permitiese diversificar la problemática del material genético y darle soluciones novedosas. Tal es el caso de la biología molecular.
Uno de sus principales objetivos es entender la relación entre la estructura de las proteínas codificadas por el ADN y su función dentro del metabolismo celular. Para ello ha desarrollado una técnica especial llamada ingeniería genética, que se basa en la manipulación directa de los genes o segmentos de ADN que codifican para determinada proteína, y de los mecanismos de expresión de estos genes. El conjunto de técnicas conocidas como ingeniería genética deriva en forma directa de la biología molecular. Veamos sus principales características.
Uno de los organismos mejor conocidos desde el punto de vista genético es la bacteria Escherichia coli. Esta bacteria es un procarionte con un cromosoma circular y genes en otras estructuras llamadas plásmidos. Como resultado de extensas investigaciones de esta bacteria en particular, se ha conocido de manera sobresaliente la estructura y función de los plásmidos. Este plásmido presenta características interesantes para su manipulación. Pero no sólo esta bacteria los presenta. Los plásmidos forman parte del material genético de casi todas las bacterias.
Son más sencillos que los virus ya que no tienen la capa proteica que caracteriza a estos últimos y no son más que moléculas circulares de ADN de doble hélice que se multiplican independientemente del resto de la célula. Los plásmidos tienen solamente entre el uno y el dos por ciento del total del material genético de la bacteria, lo cual los hace excelentes objetos de estudio y manipulación. A pesar de su pequeño tamaño se sabe que la información que contienen codifica para características importantes como la conjugación bacteriana, resistencia a sustancias tóxicas como los antibióticos, etcétera.
Estas características, y muy en especial la importancia clínica de la resistencia a los antibióticos, hizo que los biólogos moleculares pusieran atención a estas estructuras y descubrieran que pueden funcionar como excelentes vehículos para introducir genes no bacterianos a la bacteria a la cual pertenecen por un procedimiento de clonación molecular, que es la base de esta nueva genética aplicada con grandes potencialidades para la medicina, la industria, la agricultura, etcétera.
Como mencionamos arriba, los plásmidos son moléculas de ADN circulares de doble hélice que pueden adquirir genes nuevos. Al igual que el ADN cromosomal, una cadena es complementaria de la otra: A siempre se une a T, G siempre lo hace con C.
Este ADN puede ser cortado en partes específicas con la acción de sustancias que por estas características se les ha llamado enzimas de restricción. Cada enzima de restricción corta al ADN en una secuencia especial y característica. Por ejemplo, toda vez que se halle la secuencia GAATTC (y su complementaria CTTAAG), la enzima llamada EcoRI cortará en ese punto. El resultado es que podemos cortar el plásmido y obtener los trozos deseados. A la fecha se han descrito más de cien de estas enzimas. Existe otro tipo de enzimas, las ligasas, que como su nombre lo indica ligan los segmentos que se encuentran separados, reconociendo los sitios exactos. Una característica importante que es necesario mencionar antes de seguir adelante es que en todo el ADN, desde el bacteriano hasta el del hombre, existen secuencias específicas de bases nitrogenadas que son reconocidas de manera universal por estas enzimas llamadas endonucleasas (las que cortan y las que unen). Esto nos indica que podemos cortar y pegar cualquier molécula de ADN, y no sólo eso, sino también pegar secuencias complementarias con ADN procedente de organismos distintos.
Ahora pues, ya tenemos nuestro plásmido, lo cortamos y le transferimos genes procedentes de otro organismo (cortado y unido de la manera arriba descrita). A este proceso se le llama clonación. A la molécula que se obtiene de esta manera y que presenta genes propios y extraños se le considera un vehículo molecular. Por medio de la transformación este vehículo es introducido en una célula bacteriana o no bacteriana y, así, se reproduce continuamente. El resultado será que el gene que estamos introduciendo se amplificará (reproducirá) debido a la rapidez de la reproducción bacteriana, y al crecer será capaz de generar la proteína deseada utilizando el sistema genético de la célula. De esta manera, lo que antes tardaba su tiempo, ahora es posible obtenerlo más rápida y eficientemente. Esta construcción de genomas ha sido utilizada en otras áreas de la actividad humana.
¿ Cuáles han sido los principales resultados?
En la ganadería. la ingeniería genética ha logrado que algunos animales incrementen su producción de crías simplemente haciendo mejoras genéticas a través de la introducción de hormonas procedentes de otros organismos. Por ejemplo, a las vacas productoras de leche se les proporcionan hormonas de crecimiento de bovinos a través de bacterias transformadas.
En la industria alimentaria la utilización de levaduras mejoradas genéticamente para la producción de cervezas dietéticas, la producción de mejores condimentos para la comida, así como el aumento de aminoácidos presentes en ciertos alimentos, esta siendo un campo alentador de desarrollo de la biotecnología.
Con el propósito de mejorar los productos de la agricultura, la ingeniería genética trata de manipular el genoma de ciertas plantas introduciéndole, como ya hemos visto, genes de otros organismos para así incrementar la producción de ciertas sustancias. Por ejemplo, se ha encargado de la producción de cultivos resistentes a plagas, enfermedades y suelos salitrosos. Se ha intentado, aún sin éxito, introducir genes para la fijación del nitrógeno —presentes en la bacteria Rhizobium— a plantas como el maíz, para hacer innecesarios los fertilizantes artificiales, y así la planta sea capaz de transformar el nitrógeno atmosférico en sustancias orgánicas. Otro ejemplo conocido es la producción de la jitopapa, que no es más que la fusión de células procedentes de la papa y el jitomate; la planta produce en su parte aérea jitomates y en su parte subterránea, papas. Como vemos, es promisorio este campo de experimentación en el cual la biotecnología intenta demostrar que la manipulación de los genomas puede traer buenos resultados en la producción agrícola.
En la medicina, ya lo hemos mencionado, también es importante el avance de la ingeniería genética. Se han producido a gran escala proteínas importantes, como la insulina, para el tratamiento de algunas enfermedades. La insulina —proteína que transporta la glucosa del flujo sanguíneo hacia las células y que está ausente en los diabéticos— producida por los cerdos, que ha sido utilizada tradicionalmente para tratar la enfermedad de la diabetes, tenía un costo muy alto; con la utilización de la ingeniería genética, el problema del consumo de insulina y de su costosa producción parece tener una solución. Para obtener esta producción masiva se ha extraído el gene que la produce y se ha insertado en bacterias y levaduras cuya tasa de crecimiento es acelerado, por lo que la producción de esta proteína aumenta. Existen otras proteínas cuyo procedimiento es similar al de la insulina: la somatotropina (estimulante del crecimiento), el factor VIII (proteína importante en el proceso de la coagulación de la sangre, ausente en los pacientes de hemofilia), el interferón (sustancia que secretan las células como defensa contra el ataque de virus), etcétera.
Otro avance importante se refiere a la elaboración de vacunas. Se utiliza un virus conocido, al cual se le insertan genes de los diferentes anticuerpos para que el sistema inmune se defienda de los agentes infecciosos. Una de estas pocas vacunas es la que se aplica contra la malaria. También se han desarrollado vacunas contra ciertas enfermedades bacterianas; y en 1985, se elaboró la primera vacuna contra el cáncer de mamíferos (Leukocell), que previene contra el virus que causa la leucemia en los gatos. Posiblemente el campo más invadido por la ingeniería genética sea la medicina, aunque, como ya hemos visto, sus aplicaciones en otros campos van en aumento y con resultados alentadores.
Uno de los temas más apasionantes de la genética es el desarrollo. Aquí la cuestión central es que si todas las células de un organismo pluricelular tienen la misma información genética, ¿cómo es que se expresan genes diferentes en distintos tejidos para producir órganos tan disimbolos como un ojo o una pierna? Esta pregunta ha sido hecha sin mucho éxito por muchos investigadores y sólo recientemente ha aparecido evidencia que nos acerca un poco a una respuesta. El organismo que una vez más nos ha ayudado a entender un poco de este proceso es la mosca Drosophila, en la que desde hace tiempo se conocen mutantes llamados Antennapedia que, como lo indica el nombre, tienen en vez de una antena una pata en la cabeza. Estos "monstruos" nos enseñan que un pequeño cambio genético puede ocasionar un gran cambio morfológico a través de genes reguladores de otros.
La plenipotencialidad de las células
En las bacterias, estos genes se describieron desde la década de los sesenta,
cuando J. Monod y F. Jacob (véase capítulo III) descubrieron que los genes de
caminos metabólicos son regulados en forma coordinada por otro gene, un gene
regulador que inhibe o estimula la expresión de ellos. Éste es el fundamento
de la genética del desarrollo, entender cómo se lleva a cabo la expresión diferencial
de genes en diferentes tejidos. Una posible respuesta podría ser que en los
diversos tejidos las células pierden todos los genes, excepto aquellos que se
expresan en ese tejido. Esta posibilidad se eliminó en la mayoría de los tejidos
al demostrarse que las diferentes células de un organismo tienen toda la información
genética. Esto se hizo con experimentos que revirtieron el proceso de diferenciación
y demostraron que células que originalmente expresaban los genes de un tejido
podían, en ciertas condiciones, expresar los genes de otro tejido. En particular,
J. B. Gurdon demostró que núcleos (donde está todo el material hereditario,
el ADN) de células del intestino del renacuajo implantadas en citoplasmas
de óvulos no fecundados, tenían cierta probabilidad de desarrollar un sapo completo.
Estos experimentos demostraron que la diferenciación celular es una expresión
diferencial de genes y no una presencia diferencial de genes en los diversos
tejidos. También, utilizando métodos de hibridización de ADN se
ha demostrado que las células de todos los tejidos de un organismo tienen todos
los genes.
El desarrollo en la Drosophila
El desarrollo en la mosca de la fruta se inicia, como en todos los organismos,
con la fertilización. Una vez fertilizado el huevo se llevan a cabo varias divisiones
celulares que dan origen a una estructura en forma elíptica que se llama blastodermo.
La posición que guardan las diferentes células en el blastodermo define qué
tipo de estructuras de la mosca adulta generarán (Figura 27). El descubrimiento
de este tipo de estructuras, según la posición de las células se ha hecho de
diversas maneras. En una de ellas, por medio de la cirugía se eliminan del blastodermo
diversas porciones y se analiza posteriormente cómo es la morfología de la mosca
adulta. Así, por ejemplo, si se elimina la porción intermedia de la izquierda
(Figura 27), se eliminan partes del sistema nervioso central, pero si se elimina
la parte central, la mosca adulta no tendrá alas. Otra manera de saber qué partes
del blastodermo originan las diferentes estructuras de la mosca adulta es llevando
a cabo trasplantes de diferentes porciones del blastodermo para ver qué estructuras
se desarrollan.
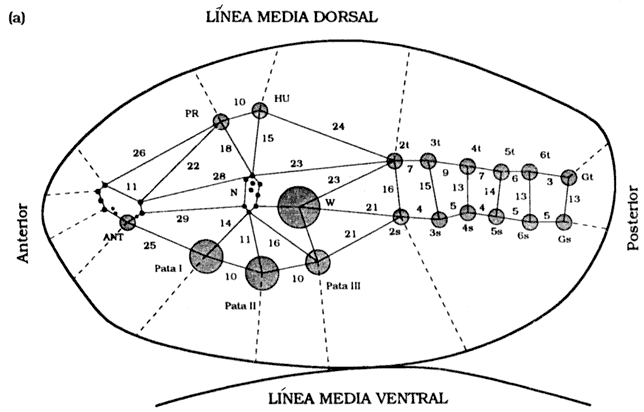
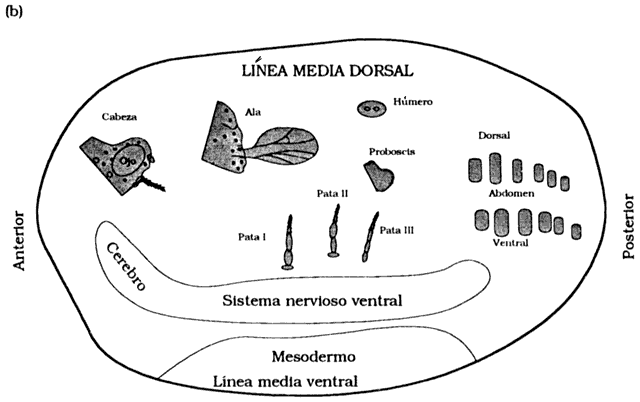
Figura 27. (A) Mapa de predeterminación del blastodermo de la D. melanogaster.
Muestra los sitios de las células que producirán las partes externas de la mosca
adulta. Las distancias se dan en sturts. Las líneas señalan las distancias
a la línea media más próxima de la mosca. El mapa se observa desde el
interior del hueco del blastodermo, hacia fuera. (b) Partes externas de la mosca
adulta representadas sobre la superficie del blastodermo. Las líneas señalan
las áreas que dan origen al sistema nervioso y al mesodermo.
Además de la mutación antennapedia que ya hemos mencionado, existen otras mutantes, llamadas homeóticas, que son mutantes de genes que tienen la capacidad de detectar la posición de las células para tomar un camino específico de desarrollo. Cuando estos genes mutan se pierde la capacidad de las células para leer correctamente la información de las posiciones. Algunas de estas mutaciones son también la ophtalmoptera, que hace que el tejido de las alas reemplace el tejido de los ojos. La mutación proboscipedia hace que la próboscis se desarrolle como una pata y la mutación cabeza tumorosa hace que el tejido de la cabeza sea reemplazado por diferentes tipos de tejido, incluyendo estructuras genitales. Quizá las mutaciones homeóticas más espectaculares sean las del complejo de genes bithorax que, como lo indica su nombre, en moscas adultas genera segmentos extras, ya que en su condición normal se produce una sustancia de desarrollo que en forma gradual modifica su concentración de la parte anterior a la posterior, de tal manera que representa una señal acerca del camino de desarrollo que pueden llevar a cabo las células según su posición. Por ello, se supone que los genes del complejo bithorax tienen la capacidad de transformar la situación posicional en expresión diferencial de genes en diferentes células.
La genética del desarrollo es, sin duda, uno de los campos que menos conocemos y del cual requerimos tener más información en el futuro. Es en ese ámbito, entre la estructura del gene, que llevó casi un siglo descubrir, y el fenotipo, que el genetista siempre ha tratado de comprender, donde se requiere investigar más en el futuro. Quisiéramos saber cómo es que de una estructura genética particular se puede expresar cierta morfología. Sólo experimentos cuidadosamente planeados y la creatividad que de vez en vez ha hecho avanzar a la genética podrán obtener esta respuesta, que es sin duda el mayor misterio que tenemos en el campo de la genética.